Jak citovat tento článek: Putzová M, Hasch M, Gomolčáková B, Šubrt I, Michal M. Výhody využití cíleného sekvenování v neinvazivním prenatálním testování. Actual Gyn. 2017;9:10-13
V současné době jsme svědky rychlého pronikání neinvazivní prenatální diagnostiky do běžné praxe. V posledních dvou letech se na trhu objevilo mnoho typů testování cfDNA jak ve formě komerčních kitů, tak v podobě “home made” testů. Cílem našeho příspěvku je upozornit na významné principiální i kvalitativní rozdíly dostupných neinvazivních prenatálních testů (NIPT) z molekulárního i klinického pohledu.
Klíčová slova: neinvazivní prenatální testování, cílené sekvenování, fetální DNA, masivní paralelní sekvenování, fetální frakce, falešná negativita, falešná pozitivita, pozitivní prediktivní hodnota, Downův syndrom, trizomie
V současné chvíli jsou dostupné v zásadě tři principiálně rozdílné typy NIPT testů. Prvním typem jsou testy založené na masivním paralelním sekvenování a následném statistickém hodnocení poměru počtu náhodných sekvencí na testovaných chromozomech v porovnání s referenčními chromozomy (BGI, Sequenom a mnoho typů tzv. „homemade“ testů). Tyto testy jsou z hlediska nákladů na provedení nejlevnější a k hodnocení dat jsou dostupné softwarové nástroje. Proto je tento typ testů tak rozšířený. Mají však některé zásadní nevýhody.
Největší z těchto nevýhod je skutečnost, že nejsou schopné spolehlivě určit zastoupení fetální frakce u všech plodů (fetální frakci u plodů mužského pohlaví lze odhadnout ze zastoupení Y-specifických sekvencí). Odhad fetální frakce je u těchto typů testů prováděn buď na základě předpokladu, že kratší fragmenty cfDNA pochází převážně z placenty, anebo na základě rozdílů v metylaci. Ani jeden z těchto přístupů však není zcela přesný. Dokonce může být zcela zavádějící, jak ukázala studie publikovaná v roce 2015 (5), kdy některé laboratoře používající tento přístup nedokázaly odlišit vzorky krve netěhotných žen od žen těhotných. Jedna z laboratoří určila míru fetální frakce u vzorků netěhotných žen okolo 4 %, což je výše fetální frakce, při které je možné provést standardní hodnocení u většiny nabízených testů. Vzhledem k tomu, že standardní a spolehlivé určení fetální frakce je považováno za základní kvalitativní parametr NIPT testování, pak jej takový test, který nedokáže odlišit pacientky s nedostatečnou fetální frakcí, nesplňuje. Výsledkem takového testu bude při nulové fetální frakci vždy normální ženský karyotyp. Do této skupiny testů patří např. NIFTY test (BGI) u nás nabízen pod komerčním názvem Prenascan, Materni T21 (Sequenom). Verifi test (Illumina) nově od 9/2016 fetální frakci uvádí, avšak metodika ani srovnání přesnosti analýzy nebyly zatím publikovány.
Použitá analýza rovněž neumožňuje bazální sledování vzorku v průběhu zpracování, záměny či kontaminace vzorku nelze při tomto způsobu analýzy odhalit.
Druhým typem testů jsou testy, které kombinují masivní paralelní sekvenování s analýzou SNPs („single nucleotide“ polymorfismů v DNA, které jsou běžné u každého jedince). Analýza SNP markerů je v těchto typech testů využívána výhradně k analýze zastoupení fetální frakce (v DNA plodu se nacházejí SNPs, které jsou odlišné od mateřských a umožňují proto spolehlivé určení zastoupení fragmentů plodu). Do této skupiny testů patří např. komerčně dostupné testy Harmony (Ariosa), nebo test Clarigo (Multiplicom). Tento přístup je k přesnému určení fetální frakce velice vhodný. K samotné analýze aneuploidií plodu však testy zařazené v této skupině opět využívají stejný algoritmus jako testy v první skupině, tedy statistické hodnocení náhodných čtení a jejich poměrů na referenčních v porovnání s analyzovanými chromozomy, stejně tak jako testy v první skupině.
Společnou nevýhodou obou těchto typů testů, kdy je hodnocení aneuploidií založeno na masivním paralelním sekvenování, je neschopnost odlišení fetálních a maternálních fragmentů sekvenovaných z mateřské plazmy. Neschopnost odlišení těchto dvou typů fragmentů a rovněž mechanizmus hodnocení vede v zásadě k několika typům falešně pozitivních a rovněž tak falešně negativních výsledků. Za zmínku stojí falešná pozitivita aneuploidií pohlavních chromozomů v důsledku maternálního mozaicizmu pohlavních chromozomů. Ta vede až k 9 % falešně pozitivních výsledků aneuploidií těchto chromozomů (22,23). Další nevýhodou tohoto přístupu je naopak falešná negativita u triploidních plodů a molárních těhotenství, které jsou tímto přístupem nerozpoznatelné. V neposlední řadě je zde riziko falešné pozitivity, případně chybného určení pohlaví plodu při syndromu mizejícího dvojčete (
Graf 1).
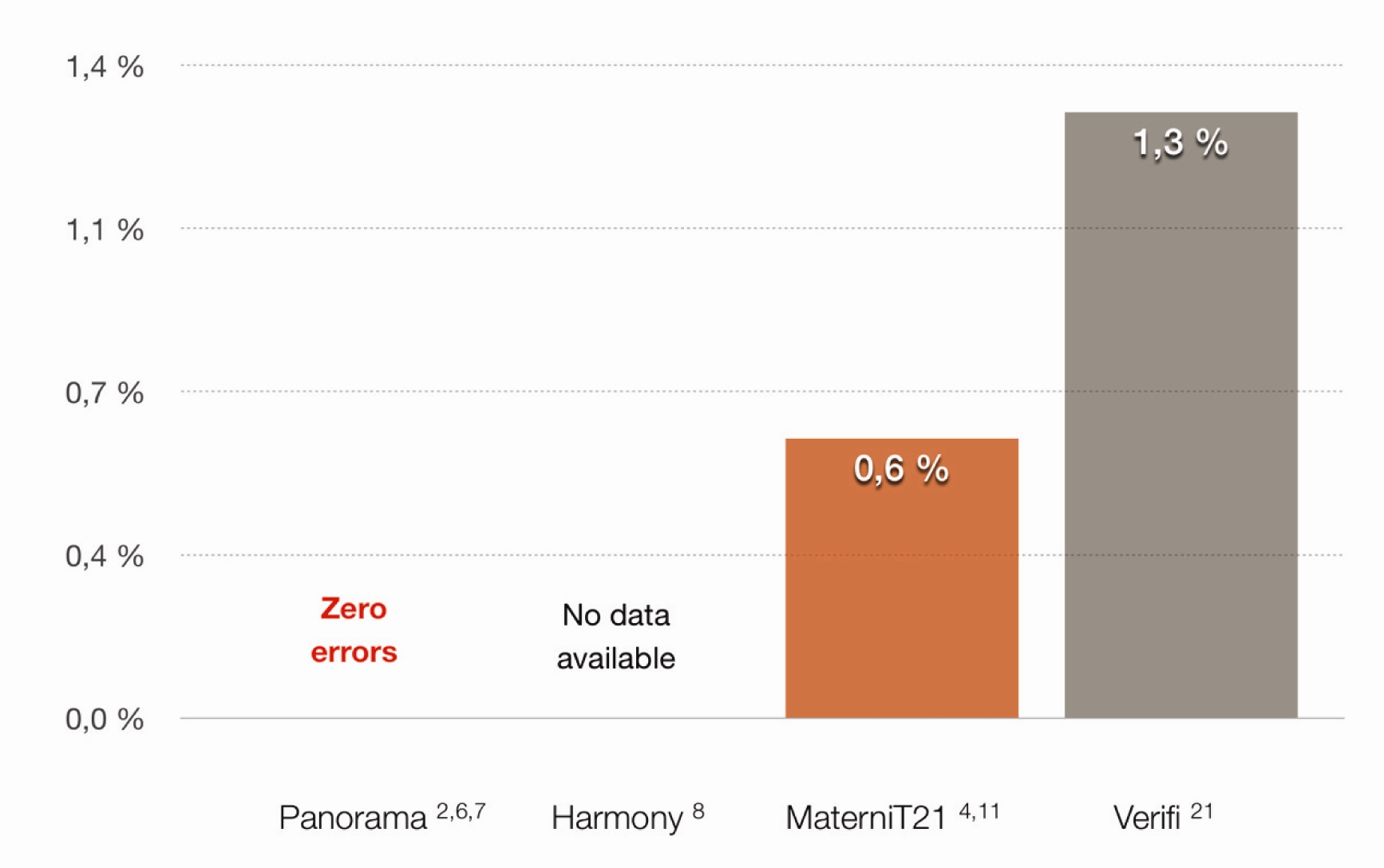
Graf 1 Porovnání spolehlivosti určení pohlaví
Třetím typem testu je test, jehož celé hodnocení je založeno na analýze SNPs. Tento test je jako jediný schopen vyhodnotit zvlášť maternální a fetální frakci v sekvenovaných fragmentech, což umožňuje snížení falešné pozitivity u maternálních mozaik a CNV’s (Copy Number Variation - delece či duplikace nekódujících či kódujících úseků chromozomů). Při publikovaném porovnání výsledků jednotlivých typů testů byla při tomto typu testu snížena FP až 7x (24) ( Graf 2).
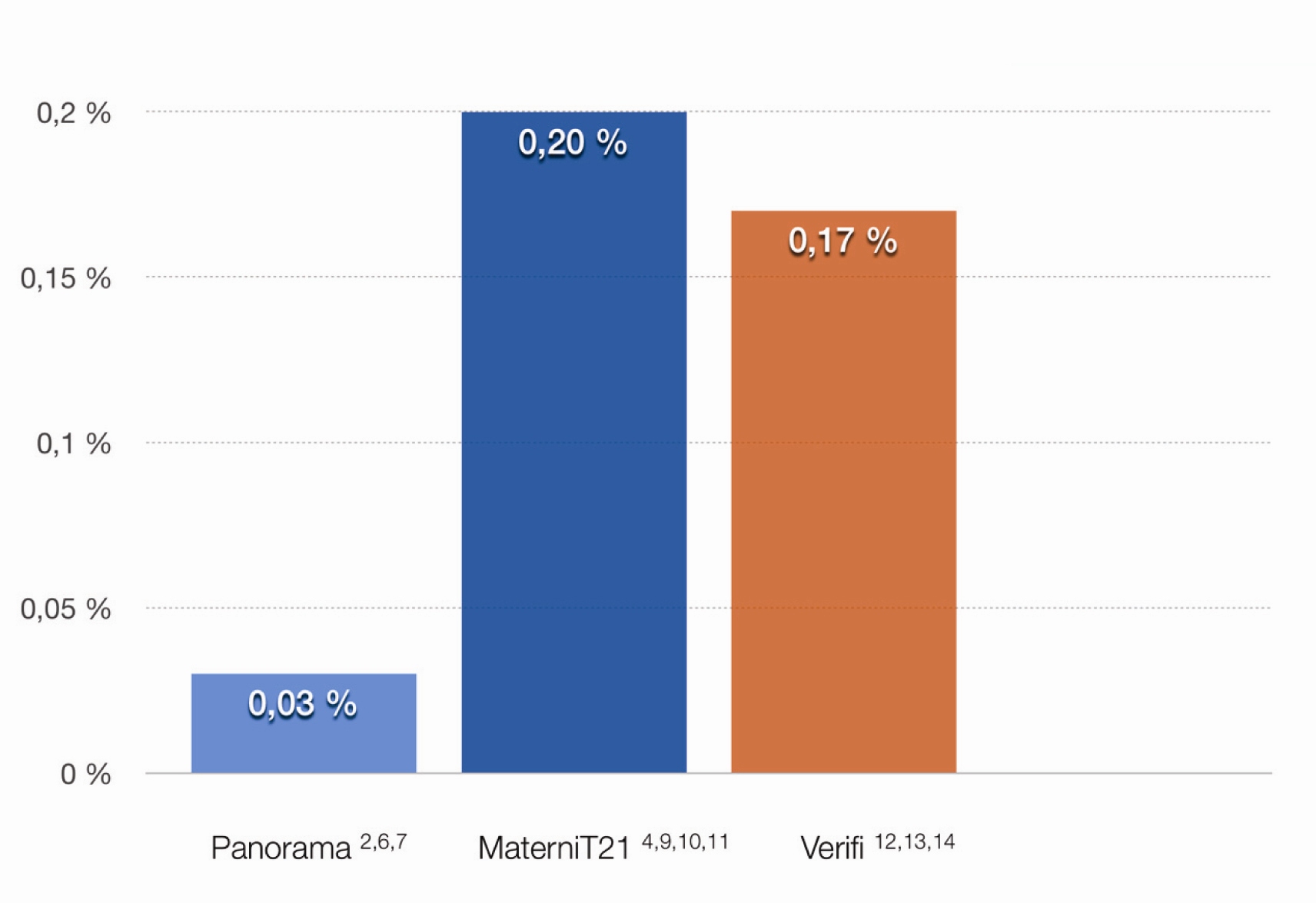
Graf 2 Porovnání falešné pozitivity (Zahrnuje FP pro trizomie 21, 18, 13 a monozomii X. Test Harmony (Ariosa) nebyl do grafu zahrnut, protože údaje o FP monozomie X nejsou v literatuře dostupné)
Tento přístup jako jediný umožňuje detekovat původ fragmentů a odlišit tak mateřskou od fetální frakce. Tím jsou podchyceny některé odchylky u plodu jako triploidie a syndrom mizejícího dvojčete, což jinými NIPT testy není možné a v konečném důsledku to vede ke snížení falešné negativity (25) (Graf 3).
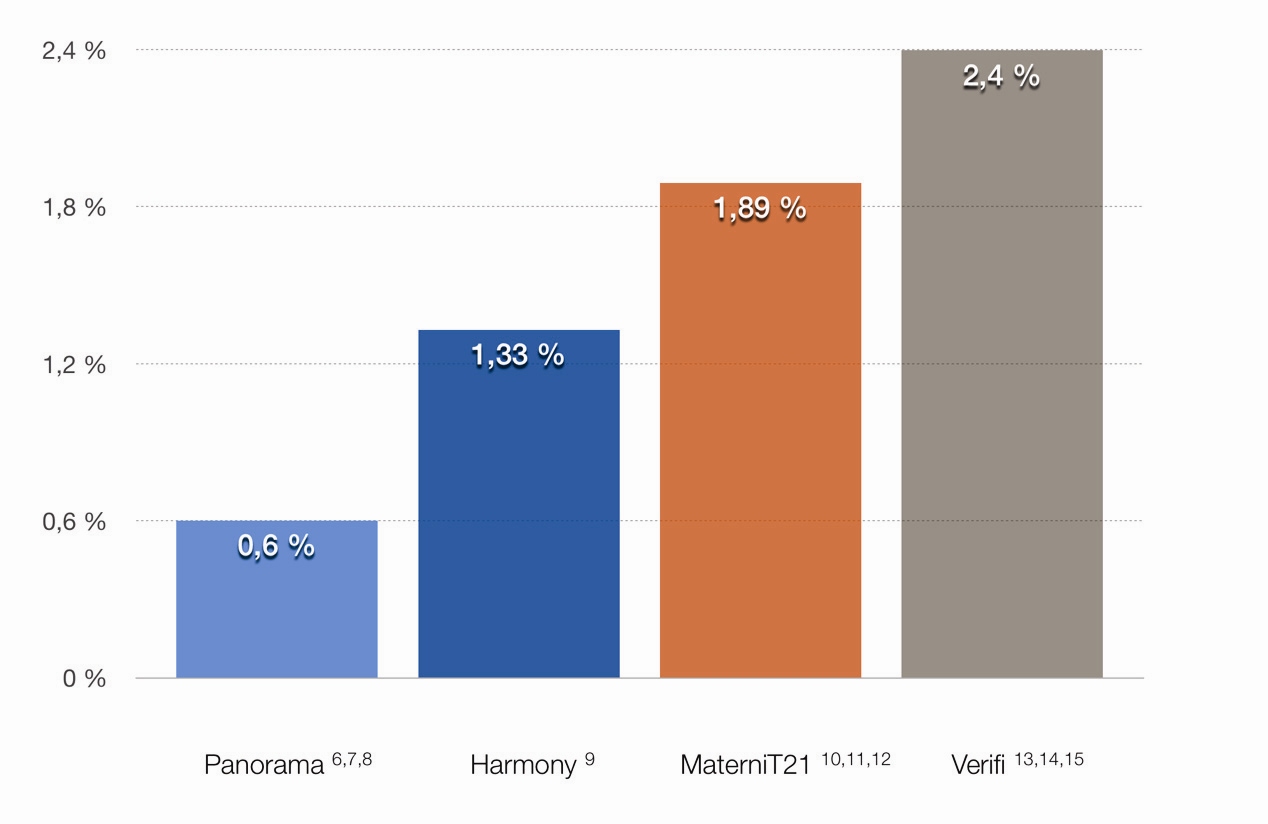
Graf 3 Porovnání falešné negativity (zahrnuje trizomie 21, 18 a 13, a monozomii X)
V současné chvíli je tento test pod komerčním názvem Panorama® firmy Natera Ltd. nabízen i v České republice, kde je prováděno i laboratorní zpracování. Bioinformatické zpracování probíhá patentovaným algoritmem s maximálními kontrolními mechanizmy: je zavedeno použití tzv. vnitřních standardů, které umožňují odhalení lidských chyb (resp. záměny vzorků) v průběhu zpracování. Způsob analýzy na bázi hodnocení SNP’s rovněž umožňuje odhalení kontaminace zpracovávaného vzorku. Všechny vzorky musí splňovat přísné kontroly kvality zpracování a musí obsahovat minimální množství 2,8 % fetální frakce, aby je bylo možné vyhodnotit. Test je validován a CE-IVD certifikován pro použití v prvním trimestru těhotenství a to od ukončeného 9. týdne. V případě, že je fetální frakce u dodaného vzorku zhodnocena jako nedostatečná (např. při vyšším BMI pacientky), je možné test později v průběhu těhotenství zopakovat. Z důvodu objektivity je nutné zmínit současně i limitace tohoto přístupu hodnocení: v tuto chvíli je vyšetření s využitím SNP’s nevhodné pro pacientky s darovanými oocyty, pro dvojčecí těhotenství a pacientky po transplantaci kostní dřeně, či časně po transfuzi krve. Tyto limitace neplatí pro metody založené pouze na masivním paralelním sekvenování, protože tyto případy nejsou použitou metodikou vůbec rozpoznány. Panorama® test je v současné chvíli pro pacientky s darovaným oocytem a dvojčecím těhotenstvím ve fázi validace. Test pro tyto indikace by měl být k dispozici v průběhu roku 2017.
Z klinického pohledu je rovněž třeba upozornit na velmi rozdílné hodnoty pozitivní prediktivní hodnoty (PPV) jednotlivých typů NIPT testů. PPV je pravděpodobnost, že osoba je skutečně nemocná, pokud je test pozitivní. Přeneseno do naší problematiky, že plod je nosičem dané aneuploidie, pokud je výsledek NIPT testu hodnocen jako pozitivní (vysoké riziko). V ambulantní praxi v případě pozitivního testu je to jeden z nejdůležitějších ukazatelů pro validní informování těhotné. PPV je závislá na incidenci onemocnění. Incidence trizomie chromozomu 21 u plodu je však rozdílná v nízkorizikové (pacientky do 35 let věku bez suspektní rodinné anamnézy (RA)) a vysokorizikové (pacientky starší než 35 let a pacientky s pozitivní RA) populaci těhotných žen. Je jasné, že pokud bude jakýkoli z testů aplikován např. na skupinu těhotných žen nad 40 let věku, bude významně nižší falešná pozitivita než u žen ve věku 20 let a tedy PPV bude poměrně vysoká. U skupiny pacientek s vysokým rizikem jsou hodnoty PPV dostupných testů bez významných rozdílů. V nízkorizikové skupině mladých žen do 29 let je však situace poněkud odlišná (Tab. 1).
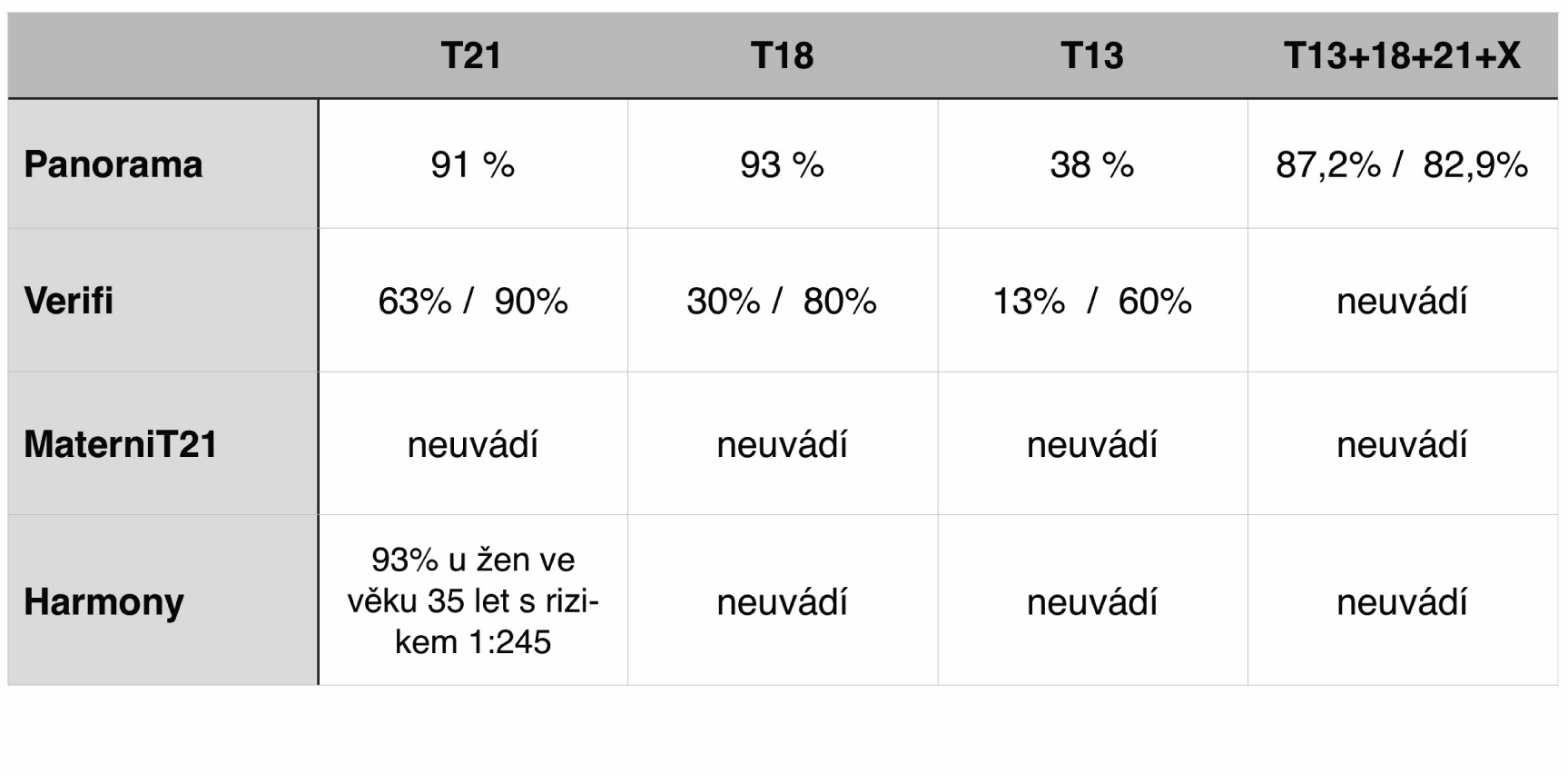
Tab. 1 PPV Low risk/high risk (interní a komerční materiály)
Pozitivní prediktivní hodnota Panorama® testu díky své unikátní technologii založené na analýze SNPs vykazuje velmi podobné hodnoty jak v nízkorizikové, tak ve vysokorizikové populaci žen, a to 87,2 % resp. 82,9 % pro celý panel aneuploidií. Pro trizomii chromozomu 21 je hodnota PPV na úrovni 91 % (26,6). V konečném důsledku to znamená dvě zásadní výhody oproti ostatním NIPT testům. Jednak je Panorama® test díky vysoké PPV i v nízkorizikové skupině nejvhodnějším testem pro celopopulační screening nejčastějších aneuploidií a jednak těhotné ženy podstupující tento NIPT test mají vyšší pravděpodobnost, že nebudou muset podstupovat invazivní test, což je základním cílem problematiky neinvazivního prenatálního testování.
Závěr
NIPT test nelze v žádném případě považovat za samospasitelný test, který by umožnil odhalit všechny chromozomální vady plodu. Dobře známé jsou biologické limitace testu: základní limitací všech typů NIPT testů je původ volné DNA, která pochází z placenty, nikoli z plodu samotného a která proto nemusí být v důsledku placentárního mozaicizmu s DNA plodu zcela shodná. Tento fakt nelze obejít žádným v současnosti dostupným testem. Na druhou stranu bývají často publikovány „limitace NIPT testování“, které jsou pouze nevýhodami použité NIPT metodiky, nikoli však obecnými limity NIPT testování. Je proto dobré znát limitace zvolené metodiky a použít ji vhodně v kombinaci s ostatními metodami prenatální diagnostiky. I v případě, že se v budoucnu stane tato metoda zlatým standardem ve screeningu nejčastějších trizomií, kvalitní USG vyšetření v I. i II. trimestru nenahradí, ale bude ho zcela jistě výborně doplňovat. Dnes již validované studie detekce mikrodelečních syndromů ukazují úžasné možnosti vyšetření cfDNA. Je to poměrně silná “zbraň” na poli prenatální diagnostiky a jako s takovou je třeba s ní umět adekvátně zacházet.
Literatura
- Canick JA, et al. The impact of maternal plasma DNA fetal fraction on next generation sequencing tests for common fetal aneuploidies. Prenatal Diagnosis. 2013;33(7):667-674, doi: 10.1002/pd.4126
- Nicolaides KH, et al. Validation of targeted sequencing of single-nucleotide polymorphisms for non- invasive prenatal detection of aneuploidy of chromosomes 13, 18, 21, X and Y. Prenat Diagn. 2013;33(6):575-9, doi: 10.1002/pd.4103
- Nicolaides KH, et al. Prenatal detection of fetal triploidy from cell-free DNA testing in maternal blood. Fetal Diagn Ther. 2014;35(3):212-7, doi: 10.1159/000355655
- Mazloom AR, et al. Noninvasive prenatal detection of sex chromosomal aneuploidies by sequencing circulating cell-free DNA from maternal plasma. Prenat Diagn. 2013;33(6):591–7, doi: 10.1002/pd.4127
- Takoudes T, Hamar B. Performance of non-invasive prenatal testing when fetal cell-free DNA is absent. Ultrasound Obstet Gynecol. 2015;45(1): 112, doi: 10.1002/uog.14715
- Pergament E, et al. Single-nucleotide polymorphism-based noninvasive prenatal screening in a high-risk and low-risk cohort. Obstetrics & Gynecology. 2014;124(2):210-218, doi: 10.1097/Aog.0000000000000363
- Ryan A, et al. Validation of an Enhanced Version of a Single- Nucleotide Polymorphism- Based Noninvasive Prenatal Test for Detection of Fetal Aneuploidies. Fetal Diagn Ther. 2016;40:219-223, doi: 10.1159/000442931
- Stokowski R, et al. Clinical performance of non- invasive prenatal testing (NIPT) using targeted cell- free DNA analysis in maternal plasma with microarraysor next generation sequencing (NGS) is consistentacross multiple controlled clinical studies. Prenat Diagn. 2015;35(12):1243-1246, doi: 10.1002/pd.4686
- Palomaki GE, et al. DNA sequencing of maternal plasma to detect Down syndrome: an international clinical validation study. Genet Med. 2011;13(11):913-20, doi: 10.1097/GIM.0b013e3182368a0e
- Palomaki GE, et al. DNA sequencing of maternal plasma reliably identifies trisomy 18 and trisomy 13 as well as Down syndrome: an international collaborative study. Genet Med. 2012;14(3):296-305, doi: 10.1038/gim.2011.73
- Porreco RP, et al. Noninvasive prenatal screening for fetal trisomies 21, 18, 13 and the common sex chromosome aneuploidies from maternal blood using massively parallel genomic sequencing of DNA. Am J Obstet Gynecol. 2014;211(4):365.e1–365. e12, doi: 10.1016/j.ajog.2014.03.042
- Sehnert AJ, et al. Optimal Detection of Fetal Chromosomal Abnormalities by Massively Parallel DNA Sequencing of Cell-Free Fetal DNA from Maternal Blood. Molecular Diagn and Gene. 2011;57(7):1042-1049, doi: 10.1373/clinchem.2011.165910
- Bianchi DW, et al. Genome-wide fetal aneuploidy detection by maternal plasma DNA sequencing. Obstet Gyneco012 May;119(5):890-901, doi: 10.1097/AOG.0b013e31824fb482
- Bianchi DW, et al. DNA Sequencing versus Standard Prenatal Aneuploidy Screening. N Engl J Med. 2014;370(9):799-808, doi: 10.1056/NEJMoa1311037
- Verifi marketing materials, 2016.
- Internal data, Natera.
- Juneau K, et al. Microarray- Based Cell-Free DNA Analysis Improves Noninvasive Prenatal Testing. Fetal Diagn Ther. 2014;36(4):282-6, doi: 10.1159/000367626
- Harmony marketing materials, 2016.
- Kim SK, et al. Determination of Fetal DNA Fraction from the Plasma of Pregnant Women using Sequence Read Counts. Prenatal Diagnosis. 2015;35(8):810–815, doi: 10.1002/pd.4615
- Sequenom marketing materials, 2016.
- Verinata white paper. Analytical validation of the Veri prenatal test. 2012
- Wang YL, et al. Maternal Mosaicism Is a Significant Contributor to Discordant Sex Chromosomal Aneuploidies Associated with Noninvasive Prenatal Testing . Clin Chem. 2014 Jan;60(1):251-9, doi: 10.1373/clinchem.2013.215145
- Snyder H L, et al. Follow- up of multiple aneuploidies and single monosomies detected by noninvasive prenatal testing: implications for management and counseling. Prenat Diagn. 2016;36(3):203–209, doi: 10.1002/pd.4778
- Benn P. Non-Invasive Prenatal Testing Using Cell Free DNA in Maternal Plasma: Recent Developments and Future Prospects. J Clin Med. 2014;3(2):537-65, doi: 10.3390/jcm3020537
- Curnow KJ, et al. Detection of triploid, molar, and vanishing twin pregnancies by a single-nucleotide polymorphismebased noninvasive prenatal test. Am J Obstet Gynecol. 2015;212(1):79. e1-79.e9, doi: 10.1016/j.ajog.2014.10.012
- Dar P, et al. Clinical experience and follow- up with large scale single-nucleotide polymorphismebased noninvasive prenatal aneuploidy testing. Am J Obstet Gynecol. 2014 Nov;211(5):527.e1-527. e17, doi: 10.1016/j.ajog.2014.08.006
- http://www.illumina.com/clinical/reproductive-genetic-health/nipt.html
- http://www.ariosadx. com/healthcare- professionals/performance/










 Rada pro výzkum, vývoj a inovace: Seznam recenzovaných neimpaktovaných časopisů vydávaných v ČR
Oficielní časopis České společnosti pro ultrazvuk v porodnictví a gynekologii.
Rada pro výzkum, vývoj a inovace: Seznam recenzovaných neimpaktovaných časopisů vydávaných v ČR
Oficielní časopis České společnosti pro ultrazvuk v porodnictví a gynekologii.



